为什么需要超多标?
简单的多色实验最多使用三种不同的荧光团,它们在可见光谱的蓝色、绿色和红色区域有不同的发射。然而,生物样本和过程的复杂性不断增加,这就在技术和应用方面提出了更高的要求,需要能在中倍数(大于 10 个荧光团)范围内同时成像的方法 [5-7]。
挑战
实现多重化的一种方法是使用上述蓝、绿、红荧光团对样品进行迭代染色和成像。然而,这一过程非常耗时,而且由于每轮染色都需要进行苛刻的处理,可能会影响样本的完整性和质量。此外,合并数据的图像处理也很麻烦,尤其是在三维组织样本中。因此,在更复杂的多重染色应用中,在一轮染色中达到所需的多重染色水平的策略已成为理想选择。虽然纯粹从样品制备的角度来看,增加样品中不同荧光团的数量是研究人员可以完成的任务,但如何处理多个荧光团激发或发射光谱的重叠问题,即使对经验丰富的研究人员来说也是一项挑战。荧光团的交叉激发和发射光谱的串扰或透射使得区分不同信号变得尤为困难。因此,样品上每增加一个荧光团,都会增加正确分离每个标签的难度。
研究方法
STELLARIS 共聚焦平台就是为这些应用而设计的。下一代 WLL 超连续激发、AOBS、优化的光束路径和多达 5 个 Power HyD 探测器同时工作,实现了超灵活的光谱能力,确保最大限度地收集来自每个相关标记的光子。 集成在 ImageCompass 用户界面中的光谱非混合工具使复杂荧光团组合的分离成为可能。
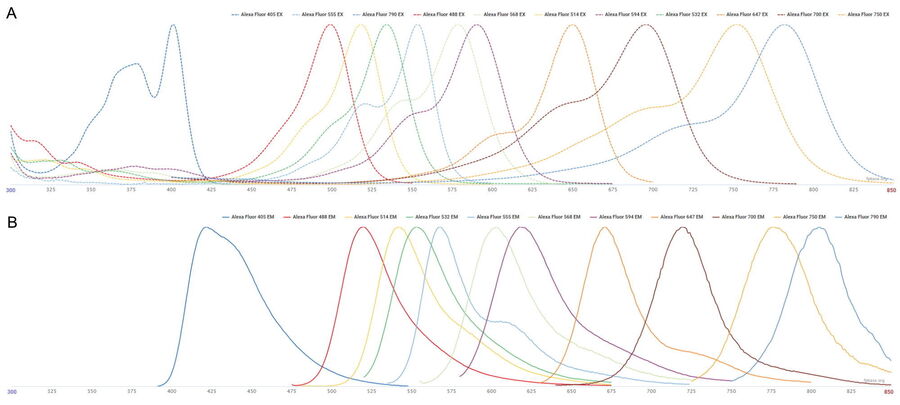
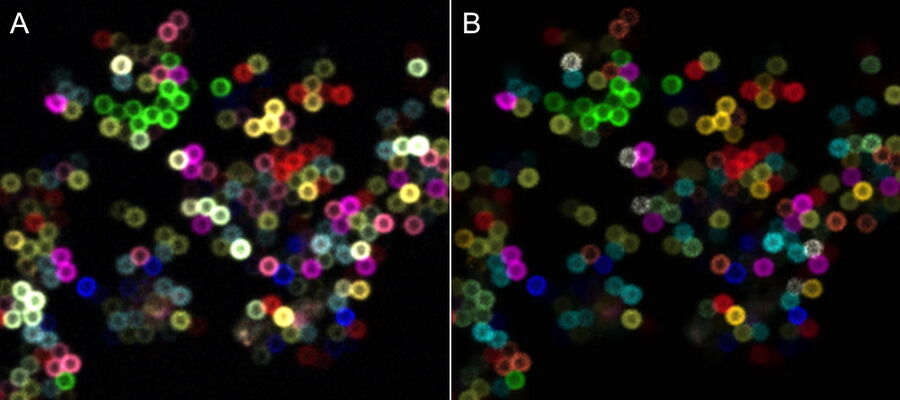
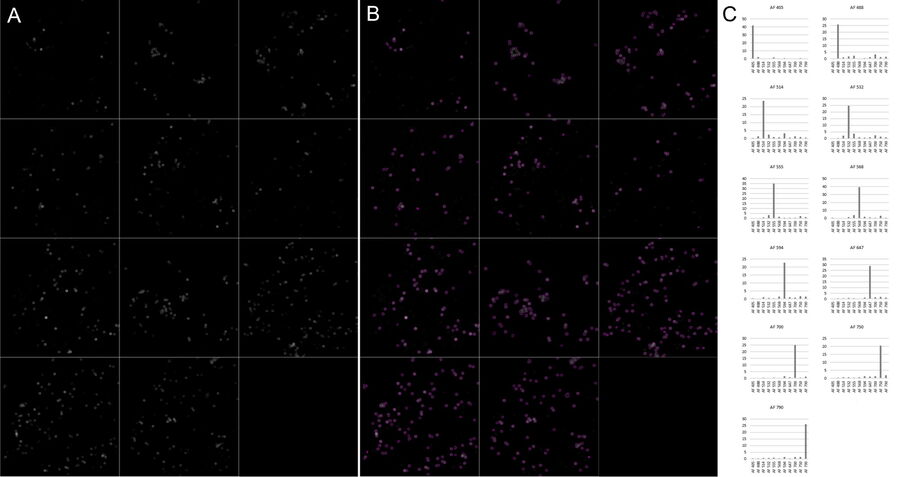
例如,我们概述了在一轮染色和成像中对 11 种不同荧光团进行成像的一种方法。我们定义了一组光谱不同的荧光团,并通过 STELLARIS 中的线性非混合通道染料分离工具将其分离。为了说明这种方法是如何工作的,我们选择了11种Alexa Fluor(AF)染料,范围从AF 405 nm到AF 790 nm(图1),与链霉亲和素连接。然后,我们将每种荧光团与涂有生物素的聚苯乙烯珠子耦合,纯化珠子,并将它们的混合物装入 Prolong Diamond 玻璃盖玻片上。使用配备了 5 Power HyD 检测器的 STELLARIS 显微镜对 11 色样品进行成像(图 2)。
图 1:本文使用的与珠子耦合的 Alexa Fluor 染料的激发光谱(A)和发射光谱(B)。光谱由 FPbase [8] (www.fpbase.org)生成。