简介
理解生物组织与材料在纳米尺度上的精细结构,不仅需要高分辨率成像,更离不开三维空间的立体语境。这正是体电子显微镜技术的不可替代之处。体电子显微镜技术(Volume-SEM)通过连续获取超薄切片的高分辨率图像序列,使研究人员能够重构细胞与亚细胞结构的精细三维模型。这一技术在细胞生物学、神经科学及癌症研究等领域具有关键意义,其核心价值体现在:
然而,即便获得了高质量的三维图像数据,如何从海量信息中提取有意义的生物学信息,仍是研究者面临的巨大挑战。这也是AI驱动的图像分割技术正在这一环节发挥革命性作用,其核心价值体现在。在本文中,我们将展示一个从生物样本包埋到AI辅助图像分割与分析的完整示范性工作流程。
结果
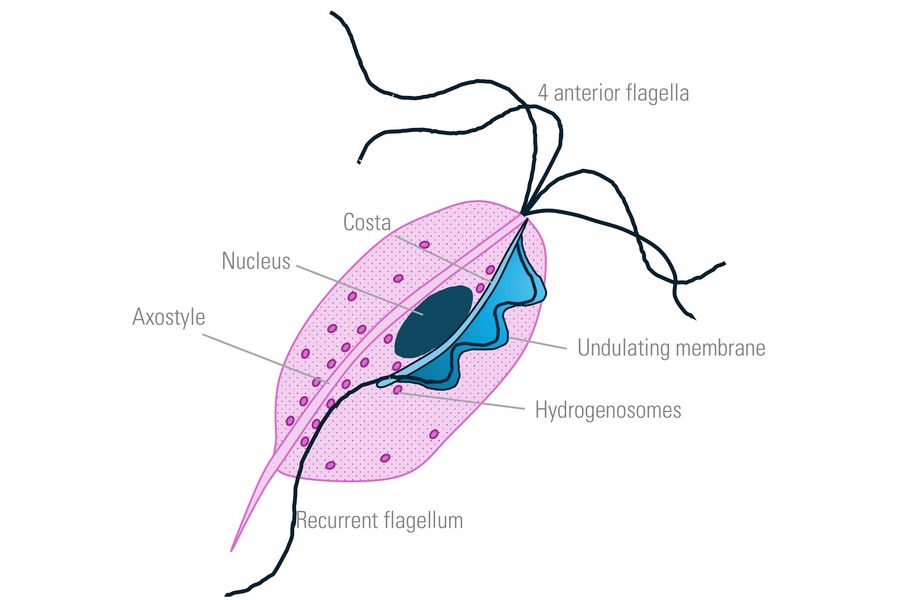
本研究选用寄生性原生动物 阴道毛滴虫 作为实验样本(如图1所示)。该生物体具有重要临床意义,它是导致滴虫病——一种全球常见的性传播感染——的病原体。该生物体呈椭圆形至梨形,为厌氧性、有鞭毛、可运动的原生动物,体长约10-20微米,具有4根前鞭毛及一片由肋条支撑的波动膜,这一特殊结构使其能够进行特有的不规则游动。
样本制备
小鼠盲肠内容物被提取后,样本在戊二醛中固定过夜,随后采用OTO法(锇酸-硫代氨基酰肼-锇酸)进行后固定与染色处理。在锇酸处理步骤中,使用亚铁氰化钾作为还原剂,以增强细胞膜结构与细胞器的对比度及区分度。后固定染色采用乙酸铀酰与天冬氨酸铅进行处理。随后,样本通过梯度乙醇系列脱水,并包埋于Araldite环氧树脂中。这种整块包埋方法能在成像过程中提供足够的对比度,无需额外的后染色处理。
修块
首先使用剃须刀片对样本树脂块进行人工预修整。随后,使用UC Enuity切片机的2D自动修块功能,自动修整出一个250 × 250微米的块面。该过程需在软件工作流程引导下,通过定义目标块面的两个对角点来完成。根据用户定义的尺寸,系统将自动执行修块操作。无论修块的深度如何,或使用何种角度的刀片(90°或45°),UC Enuity都能精确计算最优刀位,以确保达成指定的块面尺寸。
值得注意的是,徕卡显微系统的AT-4专用刀能够实现刀片在全长范围内的自动对准。这使得即使是经验较少的操作者也能成功制备出连续的切片系列。
阵列断层扫描所需的连续切片带,是通过UC Enuity上的引导式体电镜工作流程采集的(图3)。
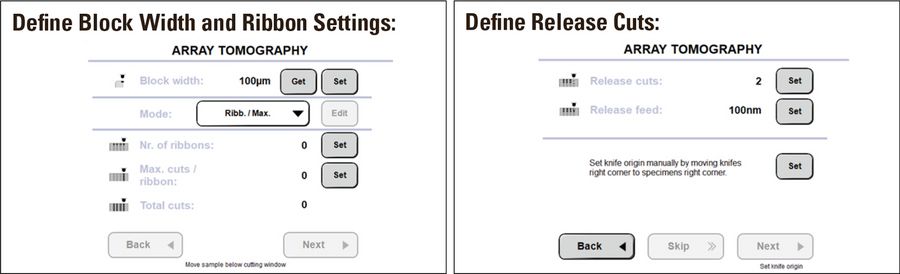
在软件工作流程中,用户将在引导下设定三个关键参数:
- 刀片上连续切片带在XY方向上的位置。
- 每条连续切片带所含的切片张数。
- 释放切片的数量与厚度
随后,切片过程便可在无需用户干预的情况下自动进行。在整个切片过程结束时,会通过切割较厚的释放切片,将连续切片带从刀片上分离并释放。这些较厚的释放切片是必要的,它们能更轻松地将连续切片带从刀锋处分离,以便将其定位于硅晶圆上。释放切片的自动生成,唯有通过电机驱动精确复刻切片过程中刀片的每一个位置才能实现。
扫描电子显微镜
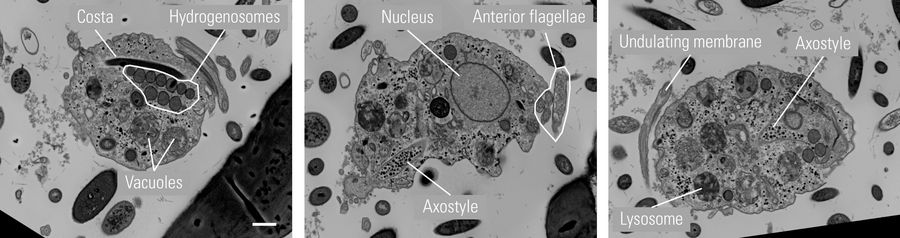
切片使用JEOL IT800SHL扫描电子显微镜进行成像,并采用专用的阵列断层扫描软件来确保切片顺序的严格遵循,以用于三维重建。在总共80张切片中,第6至64张被用于三维重建,最终形成一个总厚度为5.44微米的连续体积数据。下方展示了三张具有典型细胞器结构的示例性切片图像(图4)。
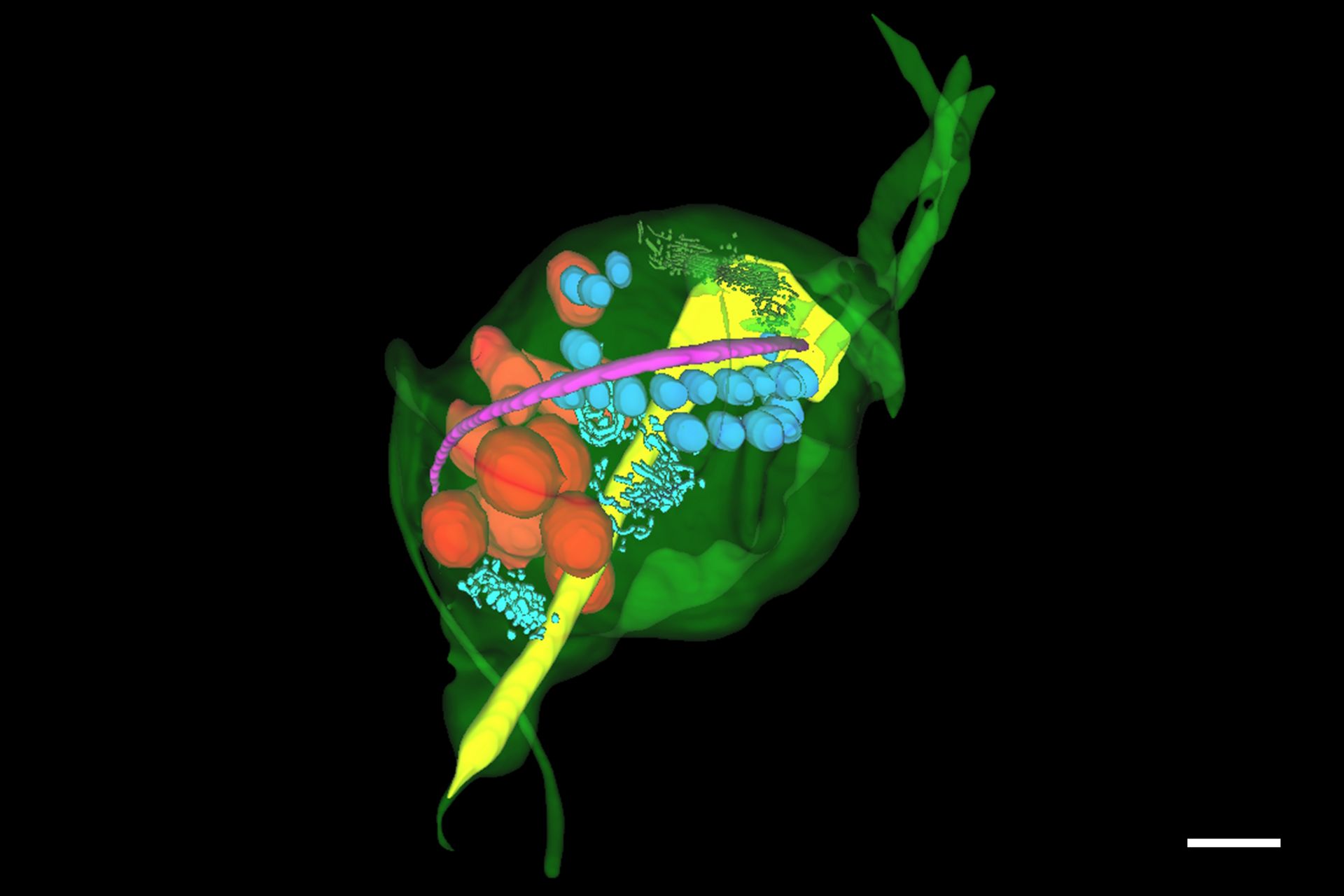
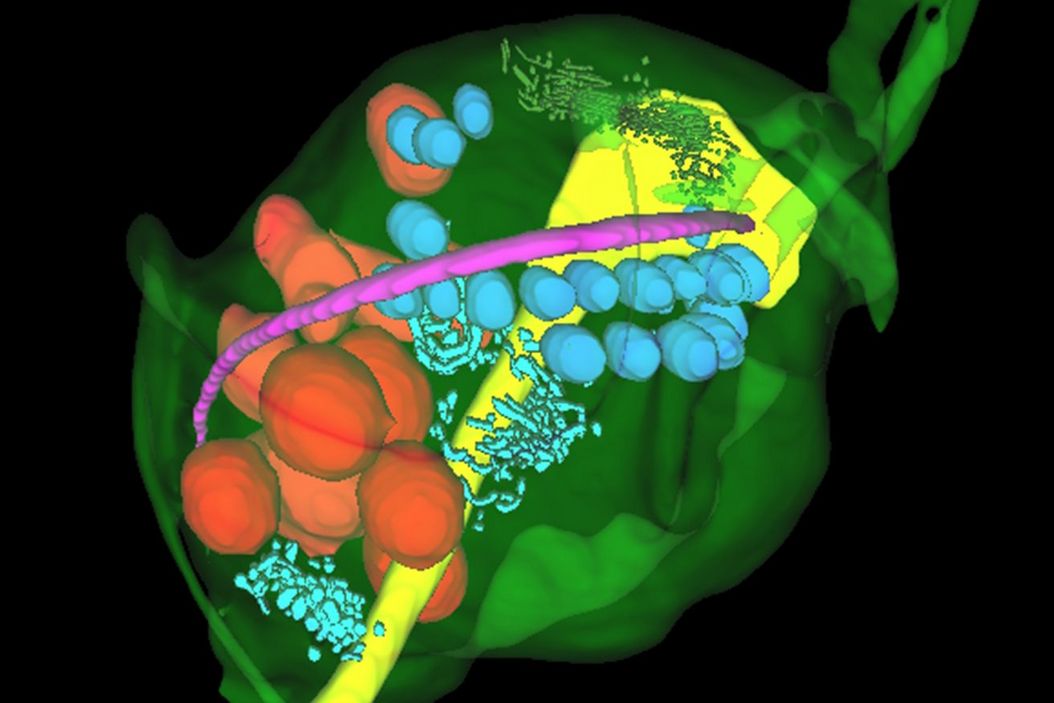
左图:肋骨突出并与起伏的薄膜相连。寄生虫的运动依赖于肋条构型的主动变化。氢化酶体是由膜包被的球形细胞器,直径约为0.5微米,通常分布于肋条附近。
中心:四根前鞭毛共同协助毛滴虫实现运动。轴柱是一种由微管构成的管状结构,从细胞核延伸至细胞后端,贯穿细胞体形成一条狭窄的通道。轴柱通过以下机制参与寄生虫的运动与细胞粘附:
右图:在原生动物中,波动膜是一种鳍状结构,能增强其在粘稠流体中的运动能力。溶酶体是由单层膜包被的球形细胞器,内含多种消化酶,负责降解细胞内废物、外来物质及受损细胞器。它们参与多种细胞分解代谢过程。
人工智能驱动的图像分割
图像采集完成后,研究人员便面临着一项艰巨的任务:在数千张连续图像中识别并标注各种结构。手动分割不仅极其耗时,还容易受到人为偏见和不一致性的影响。基于人工智能的分割算法,尤其是那些采用深度学习的算法,提供了一种强大的解决方案。
在此项研究中,我们采用了徕卡显微系统的图像分析与可视化软件Aivia,对 毛滴虫 样本的三维扫描电镜数据中的细胞器进行了分割。Aivia利用深度学习算法对二维和三维图像进行增强、分割与特征预测,从而能够更轻松地提取有意义的洞察,无需大量人工操作。
深度学习模型训练所需的真值标注
为创建用于自动检测与分割 毛滴虫 样本内不同结构(即细胞器)的“真值”数据,首先必须为其标注正确的标签。标注既可在单个Z平面(二维)上进行,也可直接在三维体数据中进行。为给深度学习模型训练提供足够信息,至少需要对两个Z平面上的所有同类型对象(即整个生物体)进行标注。
标注结果被导出为二值掩膜,并与对应的Z平面原始图像配对,作为输入文件提供给Aivia的深度学习处理器,用于训练深度学习模型。
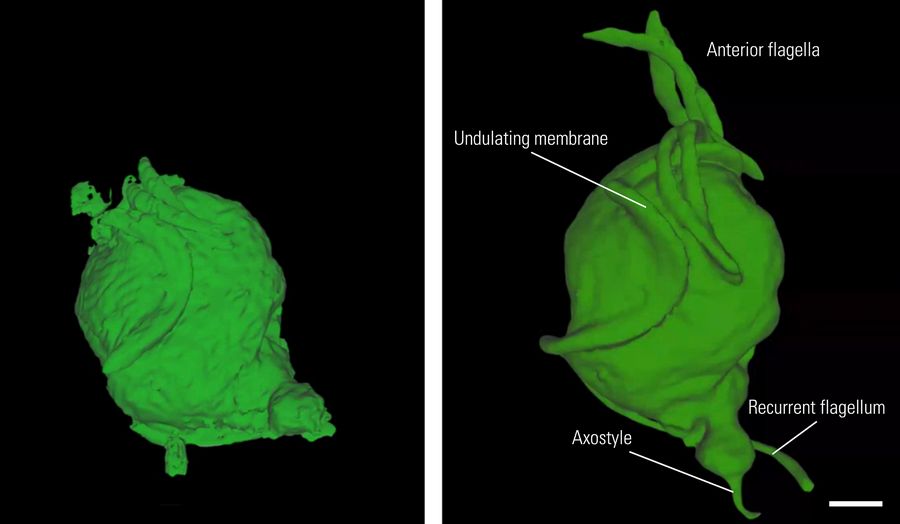
模型一经训练完成,便可重复应用于类似制备的图像堆栈,从而自动识别已学习过的结构,与对所有图像进行手动标注和分割相比,可节省宝贵时间。初步分割结果揭示了 毛滴虫 样本特有的结构特征(图5左),例如波动膜及前鞭毛的断面。
该工具利用用户在当前Z切片上对目标结构的标注,来预测该结构在下一张Z切片上的形态。通过对连续切片的逐层编辑,我们得以在初始分割结果中复原缺失的前鞭毛和向后突起的回撤鞭毛,从而重建出完整的生物体三维结构。
亚细胞结构与细胞器的三维重建与最终分割
适用于整个生物体的标注与手动编辑原则,同样可应用于亚细胞结构。视频2展示了应用预测性网格编辑技术来重建内质网的过程。
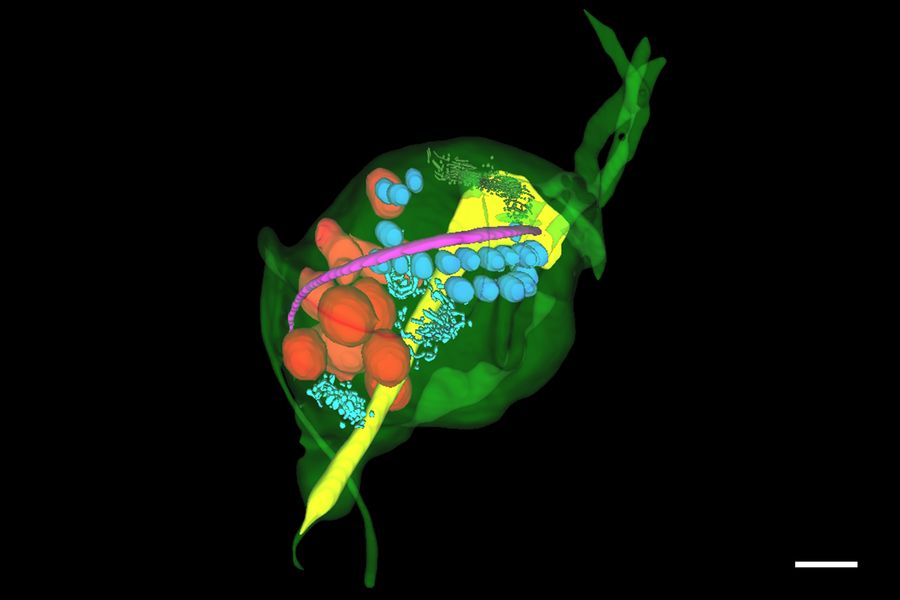
内质网独特的形态特征可被预测工具轻松识别,随后在编辑器中优化预测结果,从而实现对完整对象的快速分割。分割出的对象可被导出,用于训练新的深度学习模型,以检测图像堆栈中的其他目标结构。同样的流程被应用于重建样本中其他感兴趣的对象——包括细胞核、肋条、溶酶体、轴柱和高尔基体——最终生成了毛滴虫样本及其关键细胞器的完整三维重建模型(图6)。
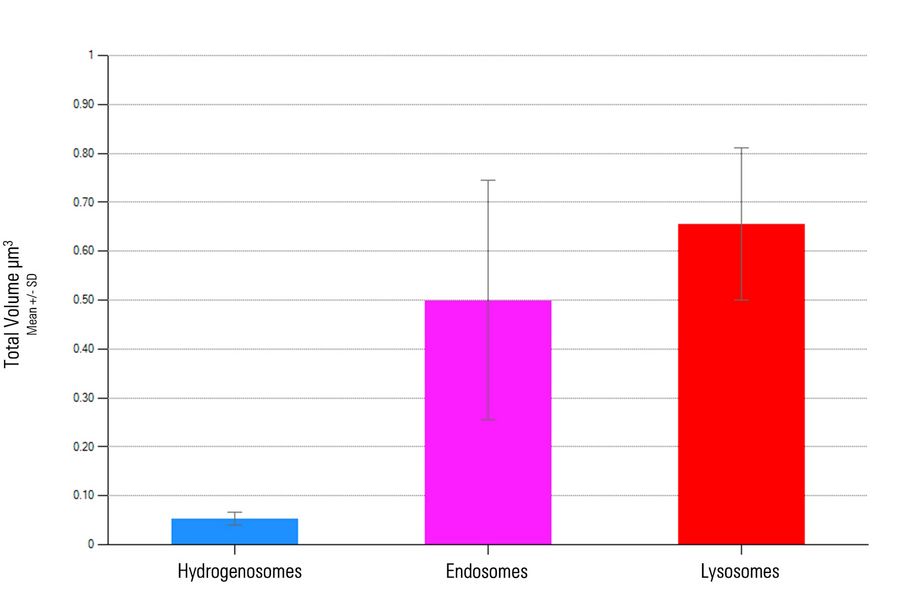
尽管氢化酶体的总体积(1.33 µm3)几乎占到内体(3.00 µm3)或溶酶体(3.28 µm3)总体积的一半,但每个氢化酶体的平均体积(0.053 µm3 ± 0.014 µm3)却显著小于单个内体(0.50 µm3 ± 0.24 µm3)和单个溶酶体(0.66 µm3 ± 0.16 µm3)。
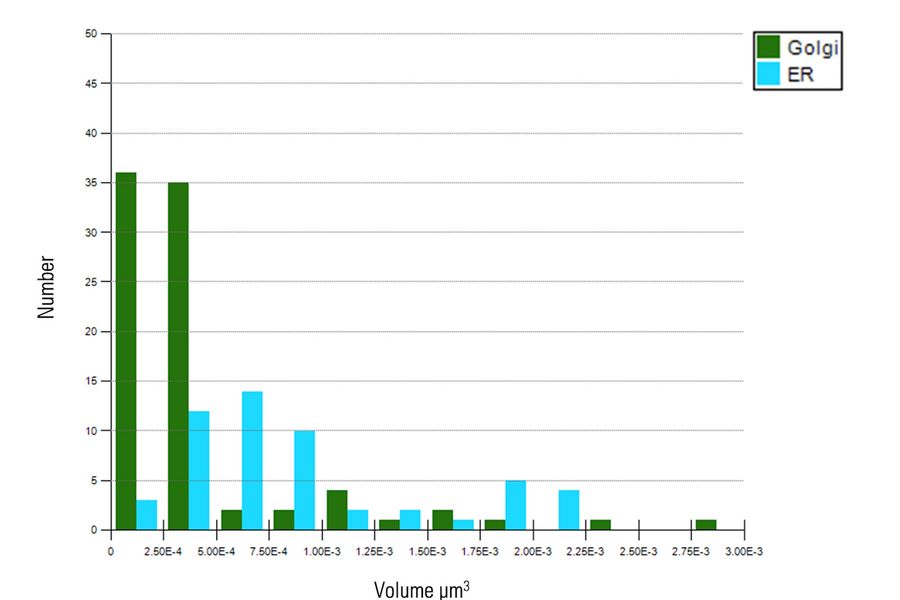
此外,我们还分析了相对较小的内质网和高尔基体片段的空间分布(图9)。二者呈现出明显不同的体积分布特征:内质网显示出更广泛的尺寸分布。通过排除较大的内质网和高尔基体结构,使得对个体对象的分析得以更有效地进行。
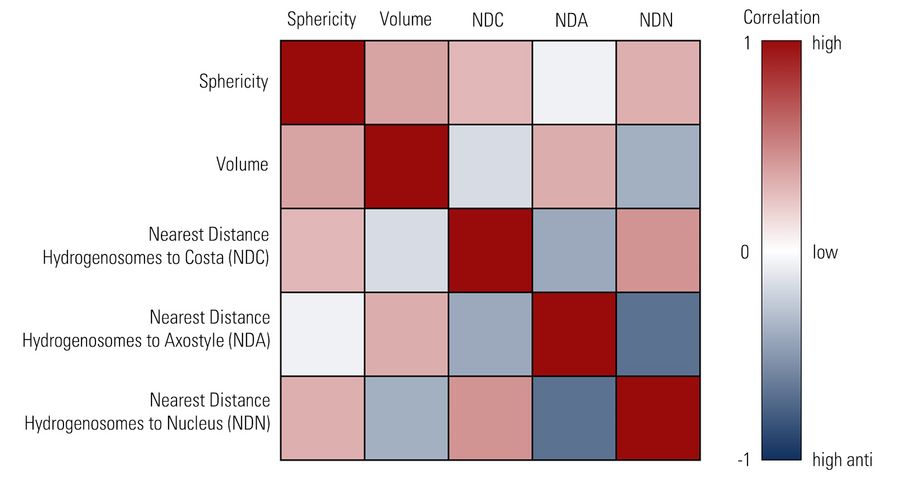
在毛滴虫体内,氢化酶体常分布于肋条附近,并通常与轴柱等其他副肋结构相关联。利用Aivia中的空间关系工具,测量了氢化酶体与其他标志性结构——肋条、轴柱和细胞核之间的距离。随后,氢化酶体形态与其到这些结构距离之间的关系被绘制成图(图10)。
总体而言,氢化酶体的大小与其到肋条的距离呈有限的负相关(r = -0.17),与细胞核的距离也呈有限的负相关(r = -0.38);而其体积与到轴柱的距离则存在微弱的正相关(r = 0.34)。鉴于氢化酶体形态已被观察到会响应氧化应激而发生改变,其形态与空间位置关系的相关性研究,可能为理解其他细胞功能紊乱提供线索。