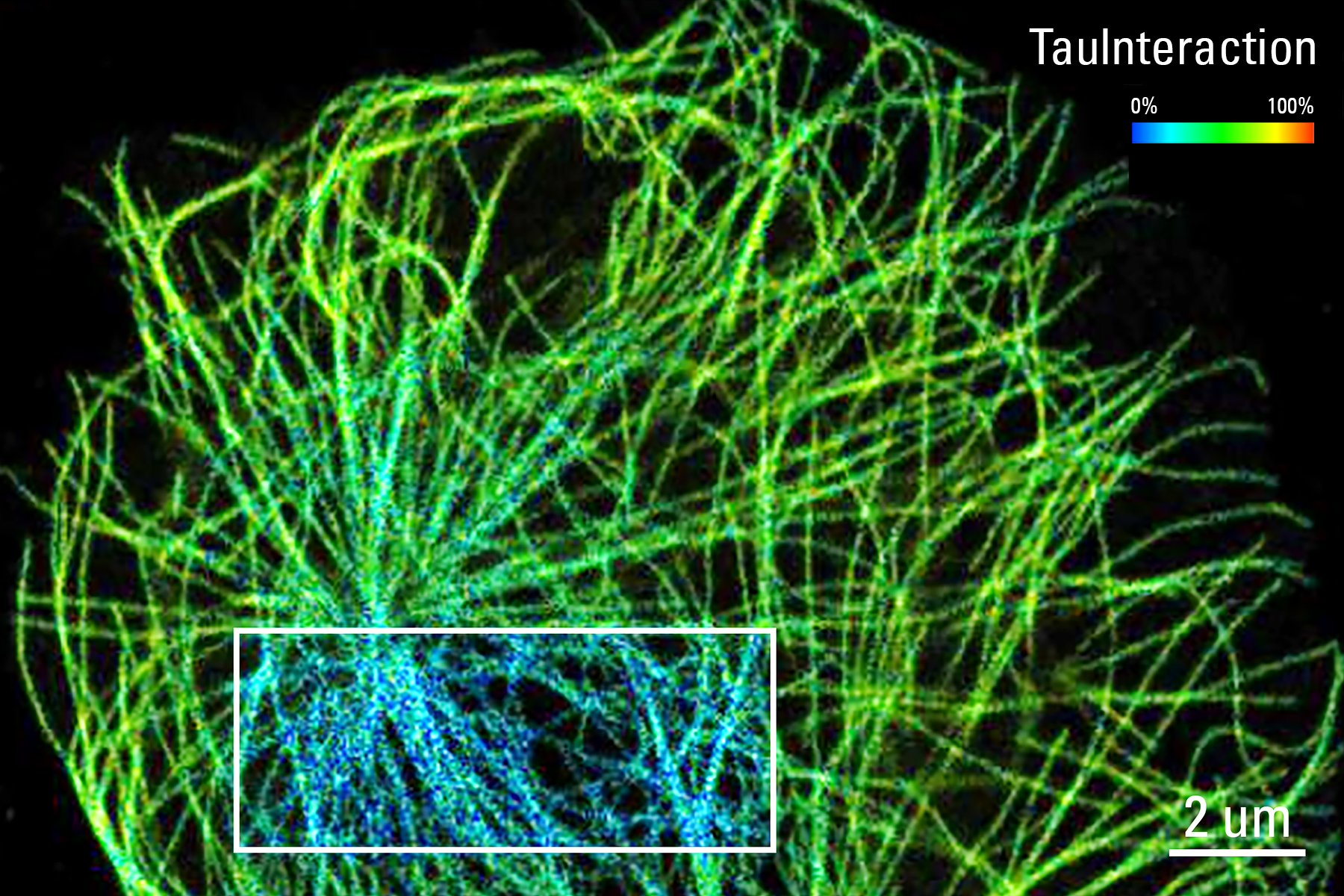
什么是TauInteraction?

分子相互作用为细胞提供能量,将基因组信息转化为细胞的各种结构(Jacob F,Monod J.1961)。广义地讲,至少有三种类型的分子相互作用:核酸(DNA/RNA)-蛋白质、蛋白质-蛋白质和小分子-蛋白质(Zhang W et al. 2002, Schlessinger K et al. 2009, Yan L and Lamb RF 2011, Dunn JA et al. 2015, Pires BRB et al. 2018, Vidya MK et al. 2018)。
通过共振能量转移(FRET,Förster 1960)实验可以确认两个分子之间发生相互作用。为了进行FRET实验,被研究的分子中的一个被标记了充当“供体”的荧光探针,而另一个分子被标记了充当“受体”的探针。这样的“供体-受体对”中供体的发射光谱和受体的激发光谱基本重叠。在这些条件下,如果两个荧光团彼此足够接近(小于10 nm,Valeur 2001)并且方向合适(Pietraszewska-Bogieland Gadella 2011)时,则可能发生FRET。距离和分子方向将决定发生了多少FRET;我们用FRET效率来衡量它,也是量化分子相互作用的最广泛的方法(Valeur 2001)。报告FRET发生状态的另一个值是相互作用供体(fD,fraction of interacting donor)的比例,它对应于在设定的时间内有多少分子参与分子相互作用(Valeur 2001)。本质上,来自供体荧光团激发态的能量通过以非辐射方式(不发射光子)转移到受体荧光团。这一过程导致1)供体的荧光强度降低,2)受体的荧光强度增加,3)供体的荧光寿命缩短(Jares-Erijman and Jovin 2006)。这些来自于供体和受体不同的信息是获取和分析FRET数据的一些策略的基础。许多FRET方法依赖于基于强度的测量,例如供体猝灭、受体光漂白和敏化发射(Berney and Danuser 2003, Padilla-Parra and Tramier 2012, Algar WR et al. 2019)。基于强度的FRET简化了仪器要求,但增加了实验设计产生假象的风险(例如,供体-受体相对信号不平衡、通道间串色、供体光漂白、光致变性)或不太适合活细胞长时间观察 (比如强光对样品带来的光毒性)。
为什么要使用TauInteraction?

FLIM(荧光寿命成像)-FRET方法仍然是FRET实验的金标准,因为FRET读数只取决于供体荧光寿命的变化,从而绕过了基于强度的方法面临的大多数风险(Padilla-Parra and Tramier 2012, Algar WR et al. 2019)。然而,FLIM实验的复杂性使得这种基于FLIM的FRET分析方法长期局限在擅长生物物理的一些科学家群体中被使用。
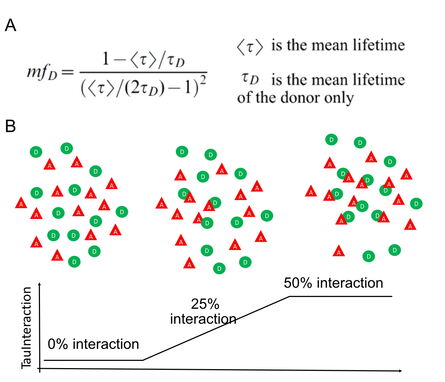
在基于荧光寿命的方法中,人们一直在努力寻找不需要复杂的物理模型,更直接的方法来探索FRET效率。这些无需拟合的方法主要侧重于提供相关数据,以便更好地理解正在研究的生物系统(Leray et al. 2013)。遵循这一理念的供体分子的最小比例(mfD,minimal fraction of donor molecules)是一个简单但很有用的概念(Padilla-Parra S et al. 2008, 2011 and Leray et al. 2013)。mfD的目的是评估参与FRET的最小分子数量。mfD与相互作用供体的比例有关(fD,Padilla-Parra S et al. 2008)。为此,mfD使用在没有受体的情况下来自供体分子的荧光寿命值参与计算(图1A)。根据这一点,它根据测量到的荧光寿命变化计算出必须与该供体相互作用的最小分子数量。通过这种方法,mfD特别适合于基于相互作用蛋白质的最小相对浓度实时地读出分子间相互作用发生的程度。
该如何使用TauSense来量化FRET?
mfD 概念非常适合于STELLARIS平台,因为STELLARIS的TauSense工具可以直接获取基于荧光寿命的信息(图一及其参考文献)。为了利用荧光寿命来定量读出FRET状态,我们实现了mfD作为一个新的TauSense工具,称为TauInteraction。TauInteraction的工作原理如下(图一):在FRET实验中,可能有许多分子(供体和受体)非常接近,如果这些是随机事件,发生FRET的平均分子数量可以忽略不计。相反,如果分子之间存在活跃的相互作用,它们保持紧密接近的时候将引起供体荧光寿命的降低。如果发生这种情况,TauInteraction会报告导致这种变化的分子比例。实验结果是一张TauInteraction图,其中包含在逐个像素级别上相互作用的分子的比例,图片将实时(即在图像采集期间)展现。此外,还提供了供体的荧光强度,如果需要,可以使用基于强度的FRET工具进行独立分析。
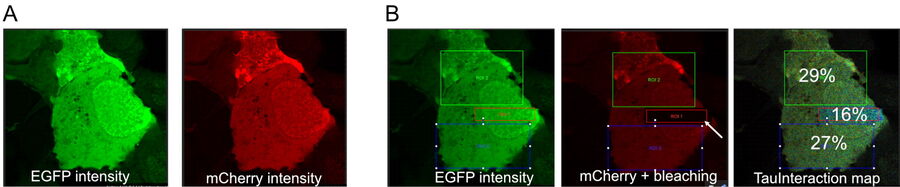
作为TauInteraction如何将分子相互作用转化为定量读数的一个例子,我们首先用串联结构来评估其表现,表达带有短氨基酸序列的蛋白质的供体-受体荧光蛋白对(EGFP-mCherry)( Tramier M et al. 2006)。这种结构在瞬时转染的活细胞中显示出恒定的正FRET行为(见图二)。有27%到29%的分子发生了互作。这与生物样本中最大可观察到的FRET是一致的(Padilla-Parra S et al. 2009)。如果我们有分离的供体和受体分子,相互作用的比例将低于这个最大值。如果我们降低受体分子的数量,我们就可以模拟这种情况。这很容易通过光漂白一些mCherry信号来实现。在光漂白一些受体并消除相互作用后,我们观察到样品中相互作用的分子的总比例下降了约50%(图二)。
在STELLARIS上集成了基于荧光寿命的工具TauSense,比如TauInteraction,为研究人员以稳定和可量化的方式研究FRET过程铺平了道路,最终目标是获得生物相互作用的功能性数据。