在EM网格上培养细胞
通常,在涂有多孔碳膜(例如 QuantifoilR)或二氧化硅(SiO2)膜的金质或钛金网格上植入急性分离或培养的细胞(图1,Mahamid等人,2019)在后续步骤中,钛金属和二氧化硅似乎更加坚硬而且稳定,无需额外添加碳层(Toro-Nahuelpan 2019)
网格通过Poly-L-Lysin或纤连蛋白(Fibronectin)实现生物激活,胰蛋白酶解离细胞在前一晚植入,以便在后续步骤中附着在碳层表面(Mahamid等人,2019)。
添加微型图案
为进入细胞样品以成功实现FIB切片并在冷冻TEM中开展后续分析,必须确保相关细胞位于网格方格的中心位置或其附近。但细胞喜欢在网格条上生长或者集簇生长,因此不适合进行FIB切片和电子透射分析。为了克服这一挑战,微型图案技术允许用户控制细胞在碳膜(图2)上的位置和分布,提高相关工作流程的可靠性。
网格表面涂有聚乙二醇(PEG),可防止生物材料附着。利用紫外激光移除该涂层,即可对细胞的黏附进行针对性控制,保证FIB切片以及TEM的可操作性(Toro-Nahuelpan 2019)。此外,可以创建特定图案,从而影响整个细胞结构并且有助于使用冷冻电子显微镜研究生物力学现象。
投入冷冻
为在固定用于电子显微镜检查的同时确保样品接近原生状态,细胞必须极速冷冻,以免产生破坏性的冰晶。这个过程称为玻璃化,因为冰片变成无结晶的玻璃状(玻璃体)
为让样品细胞达到这种效果,网格必须快速投浸到适当的冷冻剂(通常为乙烷,或者乙烷和丙烷)中。1981年,Jacques Dubochet发表了首个手动吸液和投入冷冻方法,该方法仍获广泛使用以获取出色的结果(Dubochet, J.以及McDowall, A. W.,1981)。
在投入冷冻之前,必须去除多余的液体。标准技术是使用滤纸实现受控吸液(图3,Dubochet, J等人,1982;Bellare等人,1988;Frederik, P. M.等人,1989)。
冷冻状况下的存储、装载和转移
玻璃化之后,样品必须在整个工作流程期间处于冷冻状况下。因此,必须对从存储到转移至不同成像系统的所有步骤进行冷冻处理,以免样品析晶和/或污染这尤其困难,因为这种低温冷冻样品会像磁铁一样吸引附近的湿气和灰尘。研究人员和制造商付出巨大的努力来开发并提供解决方案,以便在工作流程的不同步骤中保证样品安全。
样品通常以四个为一组存储在网格盒内,而网格盒又保存在大型液氮(LN2)罐中的Falcon多孔试管中。还可以使用更为复杂的冰球系统。
转移并装载到样品架时,通常使用液态氮(LN2)。不幸的是,LN2往往会在一段时间后,因为空气中的水分而产生结晶冰污染。在转移时,这些冰晶可能会附着到网格上,干扰随后的切片和成像过程。此外,LN2内部的能见度很低,因为它在不断移动,而且始终会有条纹。
因此,最好在LN2上部的气相部分装载并转移样品以保持冷冻条件,同时为装载步骤(图4)提供出色的可见性。
徕卡显微系统在提供GN2(气态氮)装载和转移设备方面拥有30多年的悠久历史。新的冷冻显微镜套件就在这些经验的基础上开发而成,同时融合众多客户的反馈意见打造出先进的转移舱和夹具系统。
检查样品质量和靶分布
在冷冻工作流程中,一般而言,EM操作时间尤其宝贵,因此对样品进行早期质量检查至关重要。许多因素会关系到样品能否转移到下一个工作流程步骤,包括碳箔的结构完整性、玻璃化的质量(包括冰层的厚度及其分布)、目标细胞的存在、分布和可及性,以及目标结构的存在和定位。
所有这些参数均可通过基于相机的冷冻光学显微镜(例如THUNDER Imager EM Cryo-CLEM)或使用STELLARIS冷冻共聚焦显微镜上的相机模式来检查(图5)。
透射模式显示网格、箔膜和细胞质量,反射图像显示网格表面,尤其是呈现玻璃化质量和冰层厚度,而荧光图像可以提供有关不同靶蛋白的表达水平及其分布情况的信息。
标记标志点、薄片点以及液滴中心
为了关联冷冻LM(光学显微镜)的3D图像以及后续的冷冻FIB-SEM/TEM图像,首先需要获取网格的概览图像以便大致对齐两种模式的图像(图6)。这里,反射图像非常重要,因为它们类似于SEM图像,但也可以使用透射图像。中心标记以及其他标志点(例如碳层中的缺陷)有助于快速定位并对齐概览图。
其次,需要超分辨率的共聚焦3D图像。这些图像堆栈用于在潜在薄片位置的范围内执行高精度关联。完成概览图对齐后,可以找到3D共聚焦堆栈的正确位置以便后续进行高精度关联这样做的前提是必须提供图像相对于概览图以及相对于彼此的位置。这就是Coral Cryo软件工作流程之后的处理步骤(图7)。
必须组合相机概览图像和超分辨率3D图像以检索靶区位置并在FIB-SEM上定义切片位置。这个步骤非常重要,因为在标准FIB-SEM中,无法看到荧光以及相应的靶区点位。
EM(电子显微镜)制造商近期研发出一种集成了FIB-SEM功能的荧光显微镜,可以作为在切片过程中通过检查荧光来提高工作流程的可靠性和准确性的一种绝佳选择。不过,这些系统并不具备必要的分辨率以及采集模式的灵活性,无法像单独的共聚焦系统那样实现精确的3D定位。
如何关联并检索薄片位置
作为常用的最低标准,研究人员使用LM图像的屏幕截图在EM上检索靶区的XY坐标。不幸的是,并排比较图像不仅费力耗时而且很容易出错,因此并不可靠。身为工作流程提供商,徕卡显微系统致力于通过THUNDER Imager EM Cryo-CLEM来改善这种情况。研究人员可以在图像上定位标志点和靶区标记,然后以开放EM格式的完整坐标集导出。首先,这个流程适用于2D图像,因此合乎逻辑的下一步骤就是提高分辨率并将坐标系扩展到3D坐标。
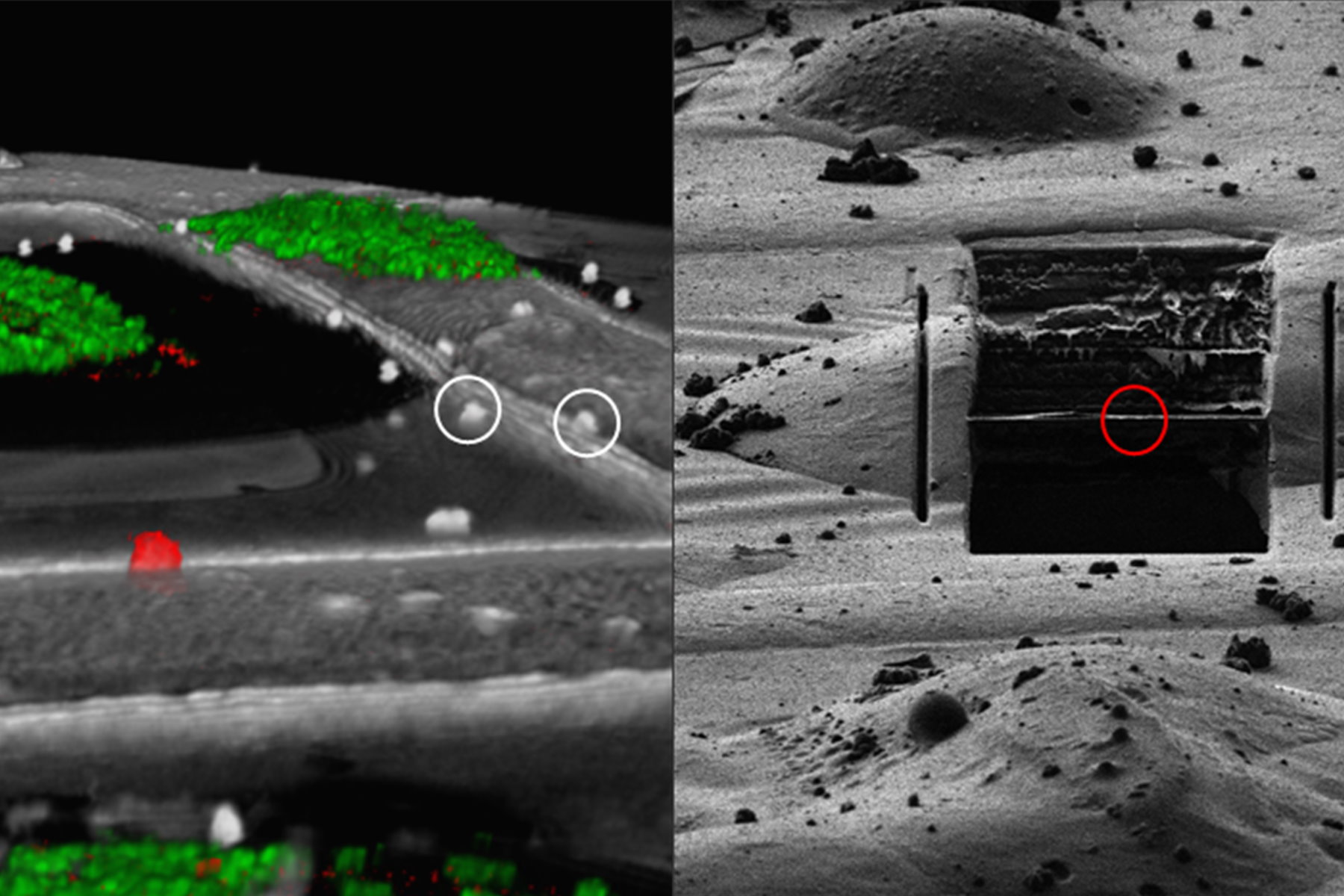
对于高精度关联和3D定位,目前广泛采用的是基于液滴的方法(Alegretti等人,2020;Klumpe等人,2021年;Bieber, A.,Capitanio, C等人,2021)液滴通常在玻璃化之前添加到细胞中,可在LM和EM中观察到,用于通过XYZ坐标对齐图像堆栈,作为图像数据相关性的基础,从而正确定位FIB切片窗口(图8)。
典型液滴的尺寸为1微米,完全呈球形,这使其中心坐标能够进行亚衍射拟合。通过SEM中的背散射电子,可以更清晰地观察到含有金属的微滴,从而将它们与大小相似的冰晶区分开来。优先选择液滴,使其荧光发射不同于实际靶的荧光发射,以便能够更好地分辨。
要使用来自冷冻LM和FIB-SEM的3D数据,在冷冻LM的引导下,进行薄片制备,可以使用一款开源软件(3D关联工具箱,简称3DCT,Jan Arnold等人,2016)。
将冷冻LM图像载入到在FIB-SEM上运行的该软件中。二维LM概览图和SEM图像之间的三点关联用于初步定位。之后,使用离子束获取相关视场,并手动点击LM堆栈和FIB图像中的相同液滴图10显示了一张LM图像和一张FIB图像,其中的靶区点位以及液滴可以在定位软件中重现其排列组合。
该软件通过对X、Y、Z信号进行高斯拟合,精准确定液滴的中心。近期的改进增加了半自动液滴检测功能以及其他功能,从而更加方便地执行冷冻FIB工作流程。(SerialFIB, Klumpes等人,2021)。
在网格条上选择围绕最终目标结构的几处液滴,作为切片处理的坐标系。基本计算方法是考虑缩放、旋转以及平移之后的线性仿射变换最后,在LM图像中选择目标结构并叠加到FIB图像上。
根据目标结构的位置,就可以定位切片窗口(图10)
Coral Cryo工作流程具有哪些优势?
Coral Cryo软件工作流程旨在为基于液滴的靶区定位工作流程提供支持。它可以提供创建合焦相机概览图像所需的成像作业(图6和图7)。所有必要的自动对焦功能均可以正确调整并分配,并且可以标记潜在薄片位置,同时能够在定义的位置执行超分辨率共聚焦Z-堆栈
在定位管理器(图11)中,可以确定所有必要的坐标标记,并且以开放格式(*.xml)提供。此类图像会自动保存,其数据格式可以导入任何FIB-SEM软件。
对齐标记用于使用相机概览图像对标记点进行初步的粗略对齐。薄片标记具有双重用途:作为进行超分辨率共聚焦3D扫描的位置标记,或者在图像采集后,作为靶结构的精确3D标记。亚像素插值确保该阶段可以在3D图像内进行高精度定位。最后,插值方法还用于标记液滴坐标,以便在FIB-SEM上进行后续液滴关联。
采用两步方法的原因在于冰污染和/或切片材料可能会沉积在薄片上。为避免在最终薄片上发生冰污染,建议采用快速抛光工艺(Schaffer M.等人,2017)。还可以采用开源的商业软件,以自动方式进行切片。
冷冻透射电子显微镜
进行冷冻FIB切片之后,含有薄片的网格转移至冷冻TEM,通过对网格(连同薄片)逐渐倾斜,采集一系列断层扫描图像。图像经过计算处理以重建所记录体积的3D断层扫描图像。通过对样品的多个图像取平均值,可以降低固有噪点,从而对蛋白质或蛋白质复合物等颗粒获得更高分辨率的结构。这种处理方式称为亚断层图像平均(Wan和Briggs,2016;Zhang 2019)。从概念上说,这相当于通过单颗粒成像(SPA),在原位实现对大分子的亚纳米分辨率。
总结
本文旨在表明冷冻共聚焦显微镜是冷冻工作流程中的一个重要组成部分,用于评估EM网格上玻璃化样品的质量和靶分布。在冷冻条件下记录的高分辨率共聚焦数据使科学家能够在3D荧光下识别目标结构。此外,3D体积可作为相关方法的参考,以便在FIB-SEM中检索靶结构进行切片,然后在冷冻TEM中进行电子断层扫描,以获得靶区的亚纳米分辨率图像。
Coral Cryo工作流程搭配新的共聚焦平台STELLARIS,再加上Coral Cryo软件,可以帮助新手用户创建网格概览图像、超分辨率3D图像以及精确的坐标标记,为后续的FIB切片和冷冻电子断层扫描奠定坚实基础。
![[Translate to chinese:] Correlation of markers in the LM and the FIB image. [Translate to chinese:] Correlation of markers in the LM and the FIB image. Correlation_of_markers_in_the_LM_and_the_FIB_image_teaser.jpg](/fileadmin/_processed_/7/f/csm_Correlation_of_markers_in_the_LM_and_the_FIB_image_teaser_52cd90c9d5.jpg)
![[Translate to chinese:] Mosaic of reflection images of a 3mm EM gold (Au) grid with holey 12 nm thick SiO2 (R 1.2/20, i.e. 1.2 μm holes with a spacing of 20 μm). HeLa cells were seeded and subsequently vitrified. [Translate to chinese:] Mosaic of reflection images of a 3mm EM gold (Au) grid with holey 12 nm thick SiO2 (R 1.2/20, i.e. 1.2 μm holes with a spacing of 20 μm). HeLa cells were seeded and subsequently vitrified.](/fileadmin/_processed_/1/1/csm_Mosaic_of_reflection_images_of_a_3mm_EM_gold_grid_ea6da0c6d9.jpg)
![[Translate to chinese:] Distribution of cells without and with micropatterning. [Translate to chinese:] Distribution of cells without and with micropatterning.](/fileadmin/_processed_/9/8/csm_Distribution_of_cells_without_and_with_micropatterning_d2a6c4337c.jpg)
![[Translate to chinese:] Controlled removal of excessive liquid by blotting prior to plunge freezing. [Translate to chinese:] Controlled removal of excessive liquid by blotting prior to plunge freezing.](/fileadmin/_processed_/2/3/csm_Controlled_removal_of_excessive_liquid_by_blotting_prior_to_plunge_freezing_0a7acd4636.jpg)
![[Translate to chinese:] Loading of grids under GN2 in the Transfer Shuttle of the Cryo- Microscopy Kit. The visibility in the shuttle under cryo-genic conditions is undisturbed. [Translate to chinese:] Loading of grids under GN2 in the Transfer Shuttle of the Cryo- Microscopy Kit. The visibility in the shuttle under cryo-genic conditions is undisturbed.](/fileadmin/_processed_/4/9/csm_Loading_of_grids_under_GN2_in_the_Transfer_Shuttle_of_the_Cryo-_Microscopy_Kit_8946f7a305.jpg)
![[Translate to chinese:] Different modes reveal the integrity of the grid and the target distribution. [Translate to chinese:] Different modes reveal the integrity of the grid and the target distribution.](/fileadmin/_processed_/9/d/csm_Different_modes_reveal_the_integrity_of_the_grid_and_the_target_distribution_b0c1783cc6.jpg)
![[Translate to chinese:] In-focus overview of the complete grid with different modes for identification of grid defects, landmarks for alignment and the target distribution. [Translate to chinese:] In-focus overview of the complete grid with different modes for identification of grid defects, landmarks for alignment and the target distribution.](/fileadmin/_processed_/0/b/csm_Overview_of_the_complete_grid_with_different_modes_for_identification_of_grid_defects_08179f49d0.jpg)
![[Translate to chinese:] Camera overview with confocal z-stacks. [Translate to chinese:] Camera overview with confocal z-stacks.](/fileadmin/_processed_/3/4/csm_Camera_overview_with_confocal_z-stacks_727ee27597.jpg)
![[Translate to chinese:] Maximum projection of 3D confocal image (left) and top view SEM image (right). [Translate to chinese:] Maximum projection of 3D confocal image (left) and top view SEM image (right).](/fileadmin/_processed_/f/a/csm_Maximum_projection_of_3D_confocal_image_and_top_view_SEM_image_3f298c52b5.jpg)
![[Translate to chinese:] Correlation of markers in the LM and the FIB image. [Translate to chinese:] Correlation of markers in the LM and the FIB image.](/fileadmin/_processed_/d/4/csm_Correlation_of_markers_in_the_LM_and_the_FIB_image_01480a2f20.jpg)
![[Translate to chinese:] Positioning of the milling windows. [Translate to chinese:] Positioning of the milling windows.](/fileadmin/_processed_/6/1/csm_Positioning_of_the_milling_windows_faf1e477b9.jpg)
![[Translate to chinese:] Coral Cryo- Software module. Landmarks, lamellae and bead markers can be defined within the software workflow. [Translate to chinese:] Coral Cryo- Software module. Landmarks, lamellae and bead markers can be defined within the software workflow.](/fileadmin/_processed_/7/9/csm_Coral_Cryo-_Software_module_403370269e.jpg)
![[Translate to chinese:] Targeted lamella ion beam view and top SEM view. [Translate to chinese:] Targeted lamella ion beam view and top SEM view.](/fileadmin/_processed_/f/0/csm_Targeted_lamella_ion_beam_view_and_top_SEM_view_e030298a8d.jpg)
![[Translate to chinese:] Water Flea Daphnia imaged by Electron Microscopy. Courtesy of Mag. Dr. Gruber, University of Vienna, Austria. [Translate to chinese:] Water Flea Daphnia imaged by Electron Microscopy. Courtesy of Mag. Dr. Gruber, University of Vienna, Austria](/fileadmin/_processed_/e/d/csm_EM_Application_Water_Flea_Daphnia_0d1b51542d.jpg)
![[Translate to chinese:] Projection of a confocal z-stack. Sum159 cells, human breast cancer cells [Translate to chinese:] Projection of a confocal z-stack. Sum159 cells, human breast cancer cells kindly provided by Ievgeniia Zagoriy, Mahamid Group, EMBL Heidelberg, Germany. Blue–Hoechst - indicates nuclei, Green–MitoTracker mitochondria, and red–Bodipy - lipid droplets](/fileadmin/_processed_/6/6/csm_Keyvisual-Cancer-cell-under-Cryo_Coral-Cryo_TechNote_dfb70c27e4.jpg)
![[Translate to chinese:] LNG-non-LNGHeLa cells. Cells kindly provided by I. Zagoriy, Mahamid Group, EMBL Heidelberg [Translate to chinese:] LNG-non-LNGHeLa cells labeled with light blue –Hoechst, Nuclei](/fileadmin/_processed_/c/6/csm_LNG-non-LNGHeLa-cells_8d365432df.jpg)