解锁结直肠癌的空间生物学洞见
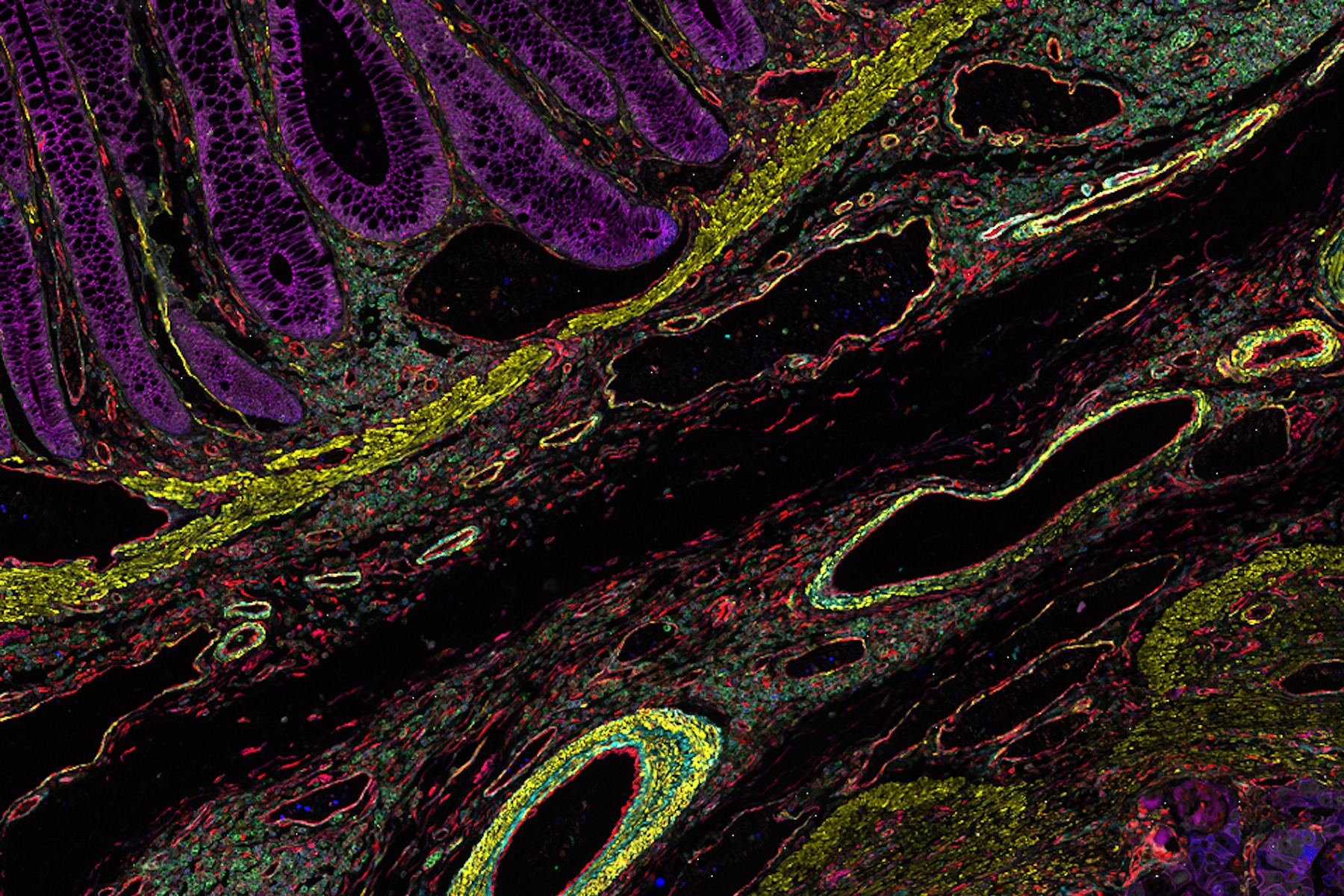
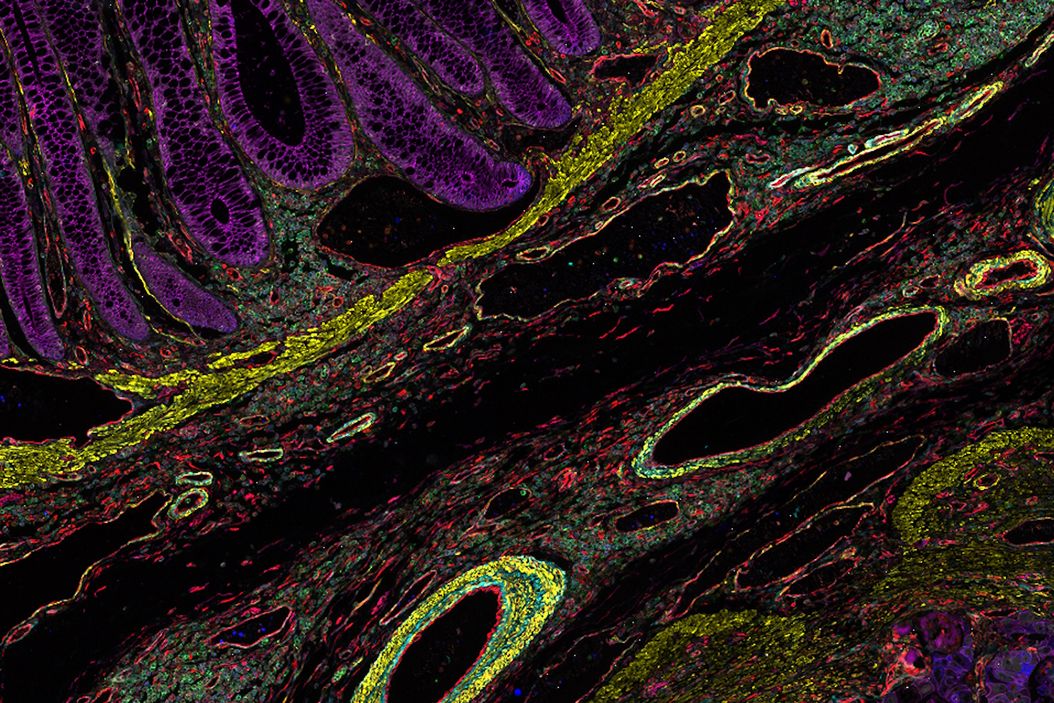
结直肠癌是重大健康挑战。虽然手术干预初期通常有效,但部分患者会复发继发性疾病且预后不良,这凸显了免疫疗法等先进治疗手段的必要性。空间生物学技术(包括 Cell DIVE 多重成像)可获取开发新疗法所需的关键见解。通过对结肠腺癌样本进行 30 种生物标志物组可视化,我们绘制了与癌症进展相关的多种生物通路图谱,包括肿瘤血管化、免疫细胞反应及细胞增殖等。AI 辅助分析揭示了细胞类型间深层次的生物学关联。该方法为理解这一重要癌症亚型的分子与结构动态提供了新视角。
通过 Minerva 探索 Cell DIVE 数据
为展示 Cell DIVE 的功能,我们提供多个可通过 Minerva 图像查看器访问的完整数据集。Minerva 是由哈佛大学科学家开发的轻量级叙事型图像浏览器,旨在简化研究人员间的组织病理学数据集的演示与共享 [1]. 该工具允许用户直接在网页浏览器中查看全分辨率 Cell DIVE 数据集。通过引导式叙述,用户可以探索不同生物学背景、定位关注区域、解读染色模式,并理解不同生物标志物的选择依据。这些叙述有助于设计 Cell DIVE 研究、评估成像方案有效性,以及制定多重成像策略。
利用 AI 与多重成像技术界定细胞边界
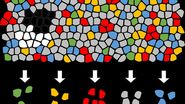
在这个 Minerva 故事中,我们识别了结肠腺癌样本中的几个关键发现,并展示了几种分析方法,这些方法可在 Aivia 等软件中使用,以充分利用空间生物学数据。科学家们经常利用图像分析软件来定义细胞边界、分析细胞表型,并测量特定细胞类型的形态和空间特征。我们使用一种称为图像分割的技术来开始提取这些信息的过程。对于这项任务,我们使用 Aivia 中提供的 2D 空间生物学工具,该工具利用基于 AI 的分割技术,提供反映实际细胞边界的高度准确的组织分割图。一旦绘制出单个细胞的边界,我们就可以利用这些区域内的相关生物标志物,利用染色阳性的生物标志物分辨特定细胞,并最终识别它们的个体表型。使用 Cell DIVE 进行多重成像可以在研究中探索更多的生物标志物,从而利用如 NaKATPase 这样的单个细胞区别生物标记物来辅助下游分析。
Minerva 图像分析平台使研究人员能够通过分析将 Cell DIVE 图像转化为定量数据。该应用不仅展现了 Cell DIVE 成像技术的潜力,更成为设计与实施多重成像研究的重要资源。
亲身体验并探索交互式数据集:
探索交互式数据集